SimpleFold : 苹果开源的轻量级蛋白质折叠预测AI模型
AI智库导航-aiguide.cc为您提供最新的AI新闻资讯和最新的AI工具推荐,在这里你可以获得用于营销的AI聊天机器人、AI在商业管理中的应用、用于数据分析的AI工具、机器学习模型、面向企业的AI解决方案、AI在商业客户服务中的应用、AI和自动化工具等。
主要介绍
SimpleFold是苹果公司研究团队开发的轻量级蛋白质折叠预测AI模型,旨在通过创新的流匹配(Flow Matching)技术降低计算成本,同时保持高精度预测能力。该模型于2025年9月正式发布,标志着蛋白质结构预测领域向高效、普惠化方向迈出重要一步。其核心目标是为科研人员提供更易获取的工具,加速药物研发和新材料探索进程。
功能特点
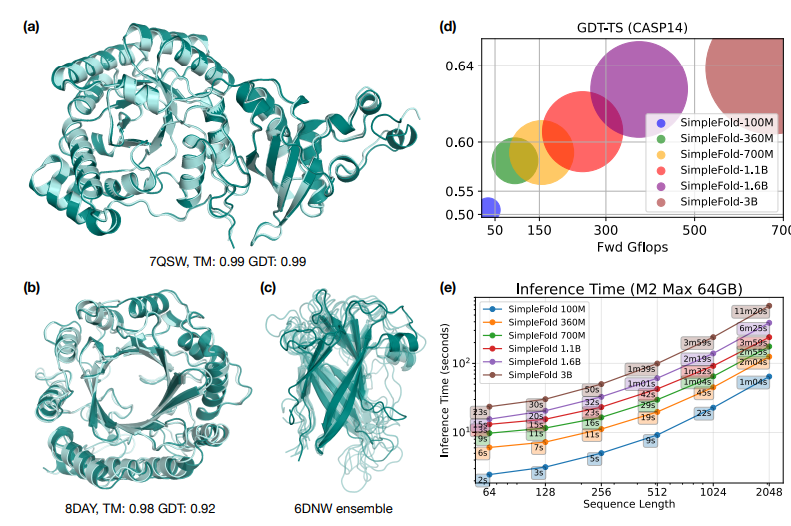
- 轻量化设计:模型参数规模覆盖1亿至30亿,支持在普通硬件上运行,降低使用门槛。
- 流匹配技术:直接从随机噪声生成蛋白质三维结构,跳过传统扩散模型的多步去噪过程,显著提升生成速度。
- 通用Transformer架构:仅依赖标准Transformer模块与自适应层,无需复杂专属设计(如多序列比对MSA或三角注意力机制)。
- 高效推理:在搭载M2 Max芯片的MacBook Pro上,处理512残基序列仅需2-3分钟,远超传统模型的小时级耗时。
- 多构象生成能力:可输出单一确定性结构或多个不同构象组成的结构集合,支持分子动力学模拟等复杂任务。
优缺点
优点:
- 计算成本低:无需依赖昂贵硬件,普通实验室即可使用。
- 生成速度快:流匹配技术使推理效率提升数倍。
- 架构通用性强:基于Transformer的模块化设计易于扩展和优化。
- 性能媲美顶尖模型:在CAMEO22基准测试中达到AlphaFold2的95%性能。
缺点:
- 对复杂结构预测仍有局限:在极端柔性或无序蛋白区域的表现略逊于专用模型。
- 训练数据依赖性:性能提升与数据规模强相关,小规模数据下可能泛化不足。
如何使用
- 访问项目平台:通过GitHub仓库或arXiv论文获取模型代码和预训练权重。
- 选择使用方式:
- 本地部署:在支持PyTorch的环境中加载模型,输入氨基酸序列即可生成结构。
- 在线推理:通过Hugging Face等平台提供的接口直接调用模型。
- 输入数据:提供蛋白质的氨基酸序列(FASTA格式)。
- 获取结果:模型输出全原子三维坐标(PDB格式)或构象集合,支持可视化工具(如PyMOL)分析。
框架技术原理
SimpleFold采用“编码器-主干-解码器”三层架构:
- 轻量原子编码器:将氨基酸序列映射为特征向量。
- 重型残基主干:基于Transformer的自注意力机制捕捉全局依赖关系。
- 轻量原子解码器:将特征向量转换为三维原子坐标。
其核心技术流匹配(Flow Matching)通过学习从随机噪声到蛋白质构象的连续变换路径,实现一步式生成。该过程由常微分方程(ODE)控制,类似“冲洗照片”般将噪声逐步转化为清晰结构,但省略了中间去噪步骤。
创新点
- 摒弃传统架构:首次完全基于通用Transformer模块构建蛋白质折叠模型,无需MSA或专用注意力机制。
- 流匹配生成范式:将结构生成视为动态轨迹积分问题,提升生成效率与质量。
- 多尺度模型设计:训练从1亿到30亿参数的多个版本,验证性能随规模扩展的稳定性。
- 对称性增强:通过SO(3)数据增强(随机旋转目标结构)使模型学习旋转不变性,提升泛化能力。
评估标准
- 基准测试性能:在CAMEO22和CASP14两大权威基准上评估泛化能力、鲁棒性及原子级精度。
- 对比实验:与AlphaFold2、RoseTTAFold2、ESMFold等模型进行性能对比,重点关注:
- pLDDT(预测置信度):衡量局部结构准确性。
- LDDT-Cα(全局距离测试):评估整体结构相似度。
- 效率指标:推理时间、内存占用及硬件兼容性。
- 多构象生成质量:在ATLAS数据集上评估生成结构集合的分布准确性与可观测性。
应用领域
- 药物研发:加速靶点蛋白结构解析,辅助小分子药物设计。
- 酶工程:预测酶的三维构象,优化催化活性。
- 材料科学:设计具有特定功能的蛋白质材料(如自组装纤维)。
- 基础研究:揭示蛋白质折叠机制,研究疾病相关突变的影响。
- 合成生物学:构建人工蛋白质,探索新型生物功能。
项目地址
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章
暂无评论...
